

全ゲノムシークエンス(WGS)など、新しい検査技術の話を聞く機会は増えたけど、自分の施設で使う日なんて本当に来るのかな?
このウェブサイトでも、これまで全ゲノムシークエンス(WGS)の海外での活用事例や、その有用性について繰り返し紹介してきました。
しかし現実的には、「WGSは研究機関や大規模な検査ラボの話であって、食品事業者には縁遠い技術」という印象を持たれている方も少なくないと思います。

確かに、WGSは比較的新しい技術であり、設備の整ったラボでは一般的になりつつあるとはいえ、食品事業者自身が導入するには、まだ多くのハードルがあります。
ただし近年、WGSそのものの技術進歩に加え、AI(人工知能)の急速な発達により、WGSは私たちが想像しているよりも早く、現場レベルで使える技術になる可能性が見えてきました。
そこで今回は、WGSが「研究室の中の技術」から「現場で使える技術」へと変わりつつある最新の状況を、海外のニュース記事をもとにご紹介します。
今回取り上げるのは、「Frontline Genomics With AI: Nanopores, Lifesaving Diagnostics, and Squiggles(AIで進化する最前線ゲノミクス:ナノポア、命を救う診断、そしてスクイグル解析)」(Global Health Now、2026年1月12日) という記事です。
「Global Health Now」は、ジョンズ・ホプキンズ大学 公衆衛生大学院が運営する、グローバルヘルス分野のデジタルメディアです。

世界中の公衆衛生ニュースを分かりやすくまとめているだけでなく、現場に根ざした独自記事も多く、非常に読みごたえがあります。



ジョンズ・ホプキンズ大学の公衆衛生大学院は、世界トップクラスの公衆衛生教育・研究機関です。私自身もニュースレターに登録し、食品安全以外の分野についても、日々多くの学びを得ています。
今回紹介する記事は医療現場の話が中心ですが、医療で先行して発展した技術は、遅れて食品安全分野にも応用されることが少なくありません(WGSも含めて)。
食品安全の実務に直接関係しないように見える記事であっても、将来の検査や監視のあり方を考えるヒントが数多く含まれています。ぜひこの機会に、最新の動向に触れてみてください。
手のひらサイズの遺伝子解析ラボ
2024年初め、ニュージーランドのウェリントン地域病院で、新生児の血液感染症が短期間に4件続けて発生しました。
通常の検査では、原因菌はいずれも「Klebsiella pneumoniae」※と判定され、特におかしな点はありませんでした。
※クレブシエラ・ニューモニエ:ヒトの常在細菌の一種。日和見感染を起こし、抗生物質耐性株の増加が医療現場で問題となっている。
しかし、同じ病棟で短期間に複数の感染が起きたこと、さらに抗生物質耐性がまったく見られなかったことから、医療スタッフは「いつもと違う」と感じ、詳しい調査を行うことにしました。
通常、全ゲノム解析(WGS)を外部の専門機関に依頼すると、検体の輸送などが必要となり、結果が出るまでに1週間程度かかります。
一方、この病院では2022年から、遺伝子解析を自施設で行う取り組みを進めていました。
Instead of waiting a week for whole-genome sequencing results from a reference lab to find out exactly what they were dealing with, staff trained by PHF employed a harmonica-size device known as a MinION genome sequencer.
(筆者訳)
通常であれば、外部の専門機関に依頼して1週間ほど待たなければならない全ゲノムシークエンスですが、訓練を受けた病院スタッフは、ハーモニカほどの大きさの「MinION(ミニオン)」という遺伝子解析装置を使って、すぐに解析を始めました。
この「MinION」は、USBメモリを一回り大きくした程度のサイズで、ノートPCに接続するだけでどこでも遺伝子解析が可能な装置です。


(画像:Oxford Nanopore Technologies)



その手軽さと高い性能を活かし、宇宙や砂漠のような厳しい環境にける遺伝子解析にも用いられています。
ナノポアと呼ばれる微小な穴を通過するDNAやRNAによる電流変化を、“うねうねした線(スクイグル)”として記録します。
このスクイグルには遺伝情報が含まれていますが、そのままでは人には理解できません。 そこで、解析ソフトがスクイグルを読み取り、DNA配列へと変換します。
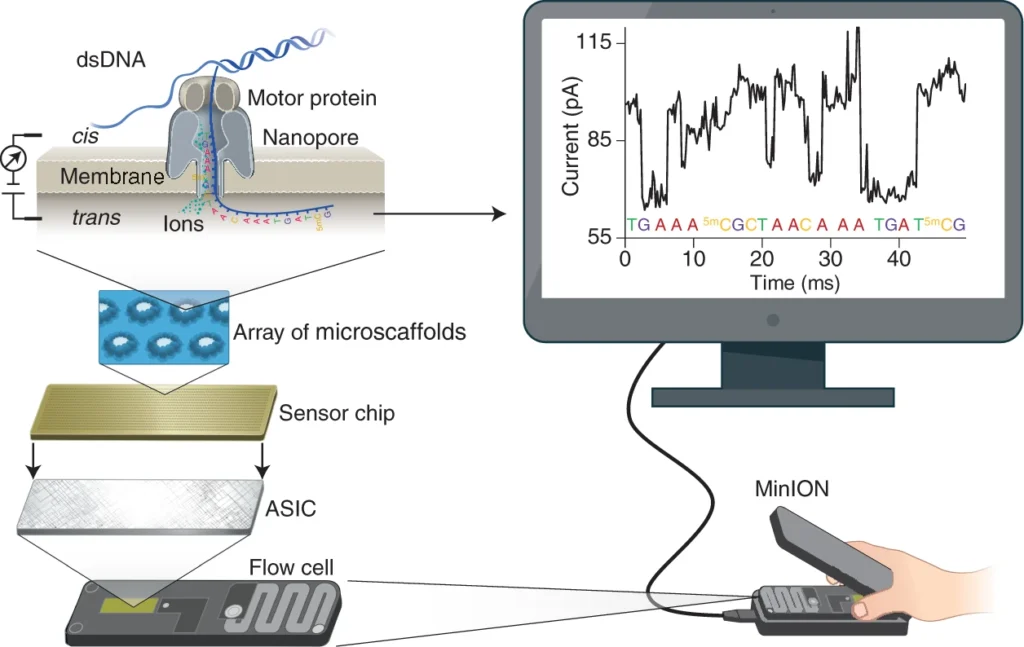

2014年にこの技術が登場した当初は、解析に12~48時間かかることもありましたが、現在では AIを活用した解析ソフトの進歩により、細菌の遺伝情報の概要を最短2~4時間で把握できる ようになりました。
In Wellington, the MinION squiggles were translated into DNA letters, revealing that three of the four isolates weren’t Klebsiella pneumoniae at all. They were Klebsiella variicola. And two of them were the same strain—ST6385—which had not been reported in clinical settings previously.
ウェリントン地域病院で、MinIONで得られたスクイグルをDNAの文字に変換したところ、4つの検体のうち3つは、当初考えられていたクレブシエラ・ニューモニエではないことが分かりました。実際には、クレブシエラ・バリイコラという別の細菌でした。さらに、そのうち2つはまったく同じタイプ(ST6385)で、このタイプはこれまで医療機関での感染例として報告されたことがありませんでした。
この結果を受け、病院スタッフは、新生児が過ごしていた部屋の環境(流し台や蛇口など)を重点的に調査しました。
その結果、新生児集中治療室の排水トラップ2か所から、同じST6385型の菌が検出されました。
流し台の消毒、共用機器の清掃ルールの見直し、手洗いなどの衛生管理強化を行ったところ、その後、新たな感染は発生しませんでした。



流し台などの環境から菌を検出し、その対策に洗浄・消毒、管理体制の見直しを行うなど、食品安全分野に通ずるものがありますね。



その通りです。それではこの記事の内容を食品安全の現場に置き換えてみましょう。
この事例は、通常の微生物検査では「よくある菌」に見えていたものが、実は別の菌・同一株による汚染だったことが、ゲノム解析によって明らかにされた例です。
つまり、従来の検査だけでは、同一汚染源による継続的な感染という「本当のリスク」を見逃す可能性があったということです。



全ゲノムシーケンスにより、詳細に調べることで初めて明らかになったということですね。
これは食品製造環境におけるリステリアやサルモネラの環境汚染をWGSで解析するのに非常によく似ています。




また、全ゲノムシークエンスのスピードも重要な要素でした。
菌を検出した際に外部機関にWGSを依頼していたのでは、どのような対応を取るべきなのかをすぐに判断をすることができません。
詳細な検査に1週間かかる場合、最悪を想定して製造ストップ、全ロット回収を行うと、問題のない製品にまで影響がおよび、多大なコストがかかる恐れがあります。
一方、迅速な解析は「疑い」を「確信」に変え、数時間〜1日以内に適切な対応の判断を可能にします。



初動の遅れが、出荷停止や回収の遅れ、被害拡大につながる食品安全の現場にとっても、医療現場と同じメリットがあると言えます。
生きているのか、死んでいるのか
PCR法は培養法に比べて感度が高く、短時間で結果が得られるため、食品分野でも培養法と並んで広く用いられる検査法となっています。
一方で、PCRには「死んだ菌のDNAも検出してしまう」という課題があります。



そのため、PCRで検出された菌が「生きているのか」「すでに死んでいるのか」が問題になることがあります。


医療や食品安全の観点で本当に重要なのは、過去に何が存在していたかではなく、今も生きていて、健康被害を引き起こす可能性があるかどうかです。
そこで現在、MinIONで得られる「スクイグル」から細菌の種類を特定するだけでなく、AIを用いて、その細菌が実際に生きているかどうかを判別しようとする研究が進められています。
In clinical settings this can be crucial. By the time a patient’s sample is sequenced, they may already have started antibiotics. The drugs may have killed the pathogen, but its DNA can still show up in the results, creating the false impression of an ongoing infection.
“If we can distinguish dead cells from live ones,” Urban says, “we avoid unnecessary changes to treatment. And if the signal shows the organism is still viable, that’s a red flag for possible antibiotic resistance and a cue to adjust therapy.”
これは医療現場では特に重要です。患者の検体を遺伝子解析する段階では、すでに抗生物質の投与が始まっていることがあります。抗生物質によって病原菌がすでに死んでいても、そのDNAを検出してしまうことがあります。すると、「まだ感染が続いている」という誤った判断を招く恐れがあるのです。
「もし、死んだ菌と生きている菌を区別できれば、本当は不要な治療変更を避けることができます。一方で、もし生きていることが分かれば、それは抗生物質が効いていない可能性があるという危険信号で、治療を見直す判断につながります」とアーバン氏は言います。
MinIONはデータの取り込みと解析をリアルタイムで行えるため、数時間、時には数分で結果を確認し始めることができます。



このリアルタイム解析により、従来のゲノム解析よりも早く、薬剤耐性の兆候をより敏感に捉えられる可能性があるとされています。
この「菌の区別」は、食品安全の現場にもそのまま当てはまります。
例えば、製品検査で病原菌が検出された場合でも、
- 生菌シグナルを検出 → 高リスク(即対応が必要)
- 死菌のみを検出 → 低リスク(追加評価で対応可能)
といった段階的なリスク評価が可能になります。
また、食品工場では、洗浄・消毒・加熱を行ってもDNAが残ることがあります。そのため、「対策が本当に効いているのか」が分かりにくい場面も少なくありません。
もし「死菌しか検出されない」のであれば、工程は有効と判断できます。一方で「生菌が残っている」場合は、洗浄・消毒・加熱の不備が示唆されます。
つまり、HACCPにおける検証(バリデーション)を、より科学的かつ実践的に行えるようになるのです。



AI×MinIONは、「速さ」と「生きているかどうか」という情報を同時に得ることを目指した、次世代の検査技術なのですね。
おわりに
全ゲノムシーケンスや AIと聞くと、どうしても「最先端すぎて自分たちには関係ない技術」という印象を持ちがちです。
しかし、今回紹介した記事から分かるように、全ゲノムシーケンスはすでに研究室の中だけの技術ではなく、現場の意思決定を支える実用的なツールになりつつあります。



装置の小型化、AIによる解析が進むことで、WGSは「特別な検査」から「必要な場面で使う選択肢の一つ」へと変わっいっているのが分かりました。
もちろん、すべての施設がすぐにこれらの技術を導入できるわけではありません。
食品安全の現場で重要なのは、最新技術を追いかけることそのものではなく、リスクを正しく見極め、早く、的確に対応することです。



「WGS」と「AI」は、そのための「新しい目」を私たちに与えてくれる技術と言えます。
「自分の施設ではまだ先の話」と感じている今こそ、「もし使えるようになったら、何が変わるのか」を考えてみることが、実現に向けた次の一歩につながると思います。









コメント