

アメリカでアイスクリームが原因のリステリア食中毒があったみたい。患者が2人しかいなくて、それぞれ別の州に住んでいたのに、どうやって2人が同じ食品が原因の食中毒だと分かったんだろう。
アメリカは面積が日本の26倍あり、とても広いです。

そして、「距離」の問題は食中毒の調査を行う上で非常に厄介です。
例えば、2023年5月にニューヨーク州でリステリアに感染した人が1人いて、さらに6月にもペンシルバニア州で1人いた場合、この2人を「同じ食品」が原因の食中毒と断定することはできるのでしょうか。
これが、同じ地域に住む2人だった場合、2人の関連性(「同じ店で食事をした」や「同じ老人ホームに入居していた」など)を見つけ、そこから共通する食品を割り出し、食中毒と断定することは可能かもしれません。
しかし、住んでいる場所が遠く離れていると、とたんに難しくなります。
アメリカではリステリアに年間1,600人ほどが感染しており、その中でこの2人にだけ共通する食品を見つけ出すことは不可能だからです。
また、患者数が多ければ、共通点(「大勢の患者がAブランドの生ハムを食べた」など)を見つけやすくなるかもしれません。
しかし、患者が2人しかいないとなるとこれも難しいです。



実はアメリカではこのような「距離」や「人数」の問題を解決する素晴らしいシステムがあります。
この記事では、アメリカでの広域な食中毒、患者数が少ない食中毒を探知するための仕組みを紹介します。
この記事を読めば、アメリカで離れた場所に住む2人のリステリア患者が、なぜ同じ食品を原因とする食中毒と断定できたのかが分かります。
PulseNetの設立
1993年に多くの州で、ハンバーガーチェーン「ジャック・イン・ザ・ボックス」を利用した人が腸管出血性大腸菌O157に感染しました。
この食中毒の原因は、加熱不十分のハンバーガーパティで、最終的に700人以上が感染し、4人の子供がなくなりました。


この事件は多くの子供が感染したこともあり、メディアの注目を集め、当時の大統領ビル・クリントンも対応に追われました。
そして、この事件はアメリカの食品安全の大きな転換点になっています。
その一つが、この事件を受け1996年にCDCが開始した「PulseNet」(パルスネット)です。
PulseNetは「検査施設間で検査結果の情報を共有するネットワーク」のことです。
PulseNetに参加している検査施設は、患者から分離された菌を同じ方法で検査し、その結果を共有します。これにより、全国どこで検査された結果であっても菌株間の類似性を比較することができます。
PulseNetでは当初「パルスフィールドゲル電気泳動 」(Pulsed-field gel electrophoresis:PFGE)という方法で菌の検査をしていました。



PulseNet の「Pulse」はパルスフィールドゲル電気泳動からきているのですね。
PFGEは、細菌のDNAを酵素で切断し、その切断パターン(バンドパターン)で菌株間の類似性を比較する方法です。
実際のPFGEの結果のイメージは下の写真のようになります。それぞれの列が患者から分離された菌です。
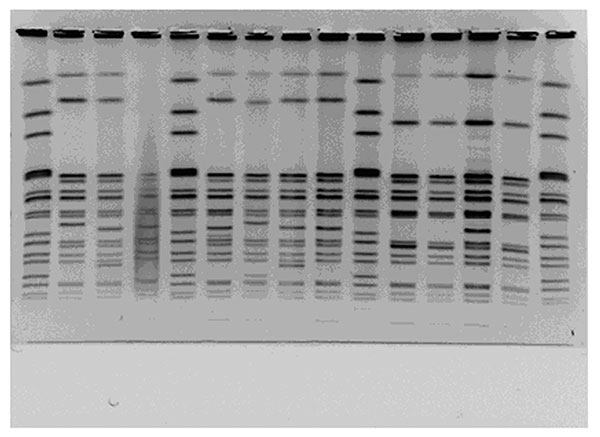

画像元:Emerging Infectious Diseases • Vol. 9, No. 4, April 2003 496-499



同じバンドパターンを持つ菌同士は、同じ食中毒の原因菌である可能性が高くなります。
PFGEがない時代には、例えばサルモネラがニューヨーク州に住む人とペンシルバニア州に住む人から検出されたとしても、この2人を同じ食品が原因の食中毒と結びつけることは困難でした。
というのも、アメリカでは年間135万人もの人がサルモネラに感染しており、2人だけに共通する食品を見つけることはほぼ不可能だからです。
しかしPFGEにより、患者が遠く離れた場所に住んでいたとしても、同じバンドパターンを持つ菌が検出されれば、同じ食品が原因の食中毒の患者である可能性が高いと考え、調査を行うことができます。
PFGEからWGEへ



PFGEでどのような食中毒でも解決できるようになったの?



PFGEにより食中毒調査の能力が格段に向上しましたが、課題もありました。
PFGEで同じバンドパターンを持つ菌が検出されたとしても、必ずしも同じ食品が原因の食中毒患者ではないことがありました。
ミネソタ州の住民37人から、同じバンドパターンを持つサルモネラが検出されました。ミネソタ州健康局はこの37人は同じ食品を原因とする食中毒だと考え調査を開始しました。
患者のうち5人はブランドBの鶏肉製品を食べていましたが、残りの32人はブランドBの鶏肉製品を食べていませんでした。
そのため、ブランドBの鶏肉製品は調査の対象から外れました。そして、最終的に健康局は原因食品を特定することができませんでした。
実は患者5人はブランドBの鶏肉製品がサルモネラに汚染されていたために起こった食中毒でした。
そして残りの32人はこの5人とは関係のない食中毒の患者でした。
(この文献を基に作成した仮定の話)
このようにPFGEでは十分に菌の類似性を区別できない場合があることが分かってきました。



これを「PFGEは解像度が低い」とも言います。
この課題を克服する新しい技術として、「全ゲノムシーケンシング」(Whole-genome sequencing:WGS)が利用されるようになりました。
簡単に言うと、食中毒菌の全塩基配列を解析し、比較する方法です。
DNAはアデニン(A)、グアニン(G)、チミン(T)、シトシン(C)が並んでおり、「AとT」、「GとC」が塩基対を構成します。ヒトでは30億塩基対、大腸菌では500万塩基対ほどあります。
このA、G、T、Cの順番をすべて解析するのがWGSです。
2つの大腸菌がいたとき、500万の塩基の順番がほとんど一緒であれば何かしら関係(例:家族や親戚)があると言えます。しかし、塩基の順番が大きく違っていれば、この2つの大腸菌は関係がない(赤の他人)と言えます。





PFGEで数個から数10のバンドパターンを見ていたのが、WGSでは500万もの情報があるため、WGSの方が解像度が高いと言えます。
2003年には「ヒトゲノム計画」でヒトの全塩基配列が解析されましたが、これには何年もの歳月と何百万ドルものコストがかかりました。これでは、WGSを食中毒調査で用いることは現実的ではありません。
しかし、2000年代初期にDNAの塩基配列を解析する技術が飛躍的に向上しました。
これにより、DNAの塩基配列の解析がより早く、そしてより安価に行えるようになり、「食中毒調査にもWGSを利用できるのではないか」と考えられるようになりました。
そこで2013年にCDCは、食中毒調査の過程で患者、食品、環境中から分離されるすべてのリステリアに対し、調査と同時進行で「PFGE」に加え「WGS」を行うという取組を試験的に行いました。
この試験的取り組みの結果、「WGSの方がPFGEより菌の類似性をより細かく区別することができること」、それに伴い「より少ない患者数でも食中毒を探知できること」が分かりました。



この取組の成功を受け、食中毒調査におけるWGSの利用は拡大しました。
2018年にはリステリアとカンピロバクターが、2019年にはサルモネラと腸管出血性大腸菌の検査がPFGEからWGSに移行しました。


WGSを使うもう一つの利点として、WGSで菌の薬剤耐性、血清型、病原性などの解析も同時に行えることです。
従来のPFGEでは、薬剤耐性などの解析を行うには別の検査を行う必要がありました。しかし、WGSで塩基配列を解析すれば、これらの情報も併せて知ることができ、迅速性、効率性が向上します。



アメリカではWGSを食中毒調査で日常的に使うことで、調査能力が革新的に向上しました。
WGSからさらに進んで
CDCが PulseNet で WGS を使い始めたのと同じタイミングで、FDAが「GenomeTrakr」という取り組みを開始しました。
GenomeTrakrもPulseNetと同様に検査機関のネットワークです。アメリカ内だけでなく、アメリカ外の検査機関も多く参加しています。
食中毒調査に限らず、人や食品、環境から分離された菌のWGSの結果と一緒に「メタデータ」もデータベースにアップロードし、共有されます。
メタデータとは、データ自体を説明するためのデータのことです。
例えば、本の場合、本の「タイトル」「著者」「出版社」「発行日」「値段」などがメタデータです。
GenomeTrakrでは、菌のWGSの結果とともに、「いつ採取されたのか」「どこで(国や地域)で採取されたのか」「何から(食品、環境、人)採取されたのか」「どんな種類の食品から採取されたのか」といったメタデータも一緒にデータベースにアップロードされます。
GenomeTrakrに共有された結果は「Pathogen Detection」でだれでも検索することができます。



GenomeTrakrのデータベースはどのように活用されるのですか?



世界中からWGSの結果と一緒にメタデータが蓄積されることで、原因食品をより早く突き止めることができるようになるかもしれません。
ニューヨーク州とメリーランド州に住む患者からサルモネラが分離されました。
患者から分離されたサルモネラの WGS の結果を GenomeTrakr で検索したところ、1件同じ WGS の結果を持つサルモネラがヒットしました。
メタデータを見たところ、インドの食品工場の環境サンプルから1か月前に検出されたサルモネラでした。
患者の聞き取り調査を行ったところ、多くの共通食があり、その中の1つにネギトロがありました。ネギトロの流通経路を調査したところ、インドで製造され、アメリカに輸入されたものでした。
GenomeTrakr により、インドの環境サンプルから同じサルモネラが検出されていたことが分かっていたため、FDAはこのネギトロを優先的に調査を行いました。
インドの製造工場にあったネギトロの検査、環境のふき取り検査を行ったところ、患者から分離されたサルモネラと同じWGSの結果を持つサルモネラが検出されました。
最終的にFDAはインドの会社で作られた「ネギトロ」が原因のサルモネラ食中毒と断定しました。
(この文献を基に作成した仮定の話)
現在のようなグローバル社会では、食品の流通は複雑で、原因食品の遡り調査は一筋縄ではいきません。さらに患者の共通食が多くあった場合、一つ一つを遡り調査するため、非常に手間と時間がかかります。
そこで、世界中の WGS の検査結果がメタデータとともに GenomeTrakr のデータベースにアップロードされることで、より迅速に原因食品にたどり着けるようになることが期待されています。
民間企業での活用



行政機関でWGSが使われていることはわかったけど、食品会社はどうなのかな?



コスト、時間、技術のハードルがありますが、食品会社でもWGSの利用は始まっています。
例えば、普段から製造環境、原材料などの微生物検査を行い、検出した場合はさらにWGSを行います。
最終製品からサルモネラを検出した場合、このサルモネラは原材料由来なのか、それとも製造環境由来なのかが解像度が高いWGSであれば、区別することができます。
そして、正確に汚染の原因が分かると、適切な対策を取ることができます。


おわりに
以上がアメリカの広域食中毒調査の仕組みの紹介でした。
この記事は「Use of Whole-Genome Sequencing for Food Safety and Public Health in the United States」を参考にしています。
アメリカのWGSの歴史がよくわかる文献ですので、気になる方は読んでみてください。
WGSの導入により、アメリカの食中毒調査の能力は革新的に向上しました。
しかし、それ単独で食中毒がすべてが解決できるわけではありません。
食中毒調査の能力を向上させるためには、WGSといった「検査」だけでなく、「患者調査」、「原因食品の遡り調査」の3つの要素の向上が必要です。
下の図のように、椅子の3つの脚をバランスよくとらないと、食中毒は解決できないということです。
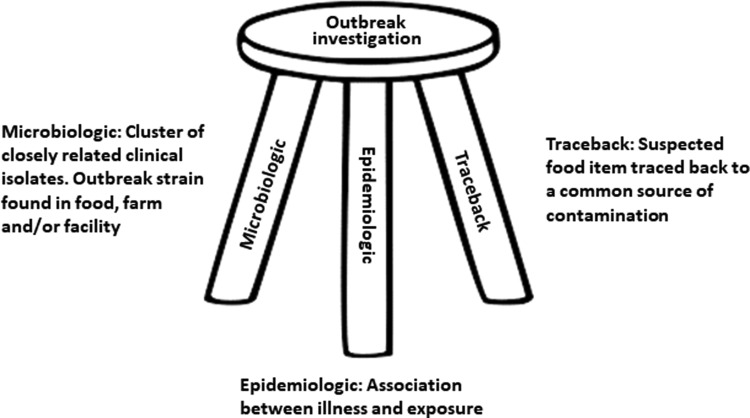




いくらWGSが素晴らしいと言っても、その結果だけに頼って食中毒がどうかを判断してしまうと、間違いが起きてしまうため、注意が必要です。
それでは日本はどうかというと、アメリカとは異なり食中毒調査で日常的にでWGSが行われていません。
そのため、今後日本の食中毒調査の能力を向上する上でWGSの導入は必要不可欠と言えます。









コメント